Mechanismus
Der Grund-Vorgang
Alle RNA-Arten einer Zelle werden durch einen im Prinzip gleichartigen Mechanismus gebildet: es wird eine Abschrift der Nukleotidfolge in einem Genabschnitt erstellt. Man bezeichnet das Umschreiben von DNA in RNA als Transkription.
Die für die Transkription notwendigen Enzyme heissen RNA-Polymerasen, oder genauer gesagt DNA-abhängige RNA-Polymerasen. Die genauere Bezeichnung ist dann nützlich, wenn man beispielsweise DNA-abhängige von RNA-abhängigen Polymerasen unterscheiden möchte. Manche Viren, die als genetisches Material RNA tragen, benötigen nämlich für ihre Vermehrung RNA-abhängige RNA-Polymerasen.
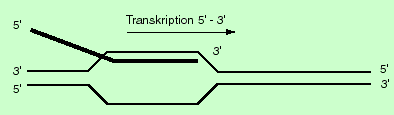
Die RNA-Polymerase kopiert die Nukleotidfolge des DNA-Matrizen-Stranges nach den Regeln der Basenpaarung. Dort, wo in der DNA ein Guanin-Baustein steht, wird in der RNA ein Cytosin-Nucleotid eingebaut, und umgekehrt. Komplementär zu einem Thymin in der DNA wird ein Adenin-Nucleotid und komplementär zu einem Adenin- ein Uracil-Nukleotid in die RNA eingebaut. Uracil nimmt also in der RNA die Stelle des Thymins ein. Die RNA-Polymerase knüpft ein Nukleotid nach dem anderen an das 3' OH-Ende einer wachsenden RNA-Kette.
Versuchen Sie zum folgenden DNA-Strang den entsprechenden RNA Strang aufzuschreiben:
3'-GCATCTGCATTCGA-5'
Lösung
X
Die Lösung sieht folgendermassen aus:5'-CGUAGACGUAAGCU-3'
Die Promotor-Operator-Region
Die RNA-Synthese muss präzise an einer Stelle in einem Gen beginnen. Es darf auch nicht irgendein Strang transkribiert werden, sondern nur der Strang, dessen Transkript die genetische Information trägt, der codogene oder Sinnesstrang. Die RNA-Polymerase (Holoenzym) bindet bevorzugt an Stellen auf der DNA, die vor einem Genanfang liegen. Eine solche Erkennungs- und Bindungsstelle nennt man Promotor.
Im Genom von E. coli sind die Nukleotidsequenzen von vielen verschiedenen Promotoren bekannt. Wenn man sie vergleicht, so fallen einige Regelmässigkeiten auf:
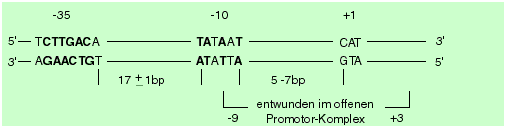
- In dem Bereich, der etwa 10 Basenpaare stromaufwärts vor dem Startpunkt der RNA-Synthese liegt (als stromaufwärts werden die Sequenzen vor dem Startpunkt, stromabwärts solche hinter dem Startpunkt, also innerhalb der transkribierten Sequenz liegende Sequenzen bezeichnet), kommt eine Sequenz von Nukleotiden vor, die eine mehr oder weniger grosse Ähnlichkeit mit der Folge 5'-TATAAT-3' hat. Diese Region wird als Pribnow-Box (nach ihrem Entdecker), als TATA-Box oder einfach als -10-Region bezeichnet (wobei als +1 das Nukleotid am Startpunkt der RNA-Synthese angegeben wird, also das erste Nukleotid der RNA).
- In dem Bereich, der etwa 35 Nukleotide stromaufwärts vom Start liegt (also in der -35 Region), gibt es innerhalb eines AT-reichen Abschnitts eine zweite Folge von oft vorkommenden Nukleotiden, häufig 5'-TTGACA-3'
 KnippersR.:
KnippersR.:"Molekulare Genetik",
6. neubearbeitete Auflage 1996,
Georg Thieme Verlag Stuttgart - New York,
ISBN 3-13-477006-7
Die Abbildung zeigt einen Muster-Promotor. Man spricht von einer Konsensus-Sequenz, weil die meisten natürlich vorkommenden Promotoren im Genom von E. coli in mehreren Positionen mit der Mustersequenz übereinstimmen. Es stimmt jedoch fast kein Promotor genau mit der Konsensus-Sequenz überein. Natürliche Abweichungen von der Konsensus-Sequenz beeinträchtigen die Effizienz der RNA-Polymerase-Bindung und der Transkriptionsinitiation.
Die -35- und -10-Sequenzen werden durch DNA-Abschnitte, die stromaufwärts oder stromabwärts liegen, beeinflusst. Diese Regionen, welche entweder vor der -35 Region oder aber nach dem Startpunkt liegen, nennt man Operator.
Initiation
Die Initiation der Transcription ist ein wichtiger Angriffspunkt für die Kontrolle der Genexpression. Häufig ist die Entscheidung darüber, ob an einem bestimmten Promotor initiiert wird oder nicht, der wichtigste oder gar einzige Schritt bei der Festlegung, ob ein Gen ausgeprägt werden soll. Wie wird nun die Fähigkeit der RNA Polymerase, an einem bestimmten Promotor zu initiieren, kontrolliert?
Wir teilen zunächst die Promotoren in zwei Klassen ein:
- Einige Promotoren können vom RNA-Polymerase-Holoenzym allein erkannt werden; in diesen Fällen wird ein zugänglicher Promotor immer transkribiert. Die Verfügbarkeit eines Promotors wird durch Bindung von Proteinen festgelegt. Diese wirken entweder direkt am Promotor, indem sie der RNA Polymerase den Zugang versperren, oder indirekt über die Kontrolle der Genomstruktur in dem betreffenden Bereich.
- Andere Promotoren können die Transkription alleine nicht angemessen unterstützen; es werden zusätzliche Proteinfaktoren benötigt, damit die Initiation stattfinden kann. Diese Proteinfaktoren wirken gewöhnlich, indem sie DNA Sequenzen erkennen, die sich in der Nähe der von der RNA-Polymerase gebundenen Sequenz befinden oder sogar mit dieser überlappen.
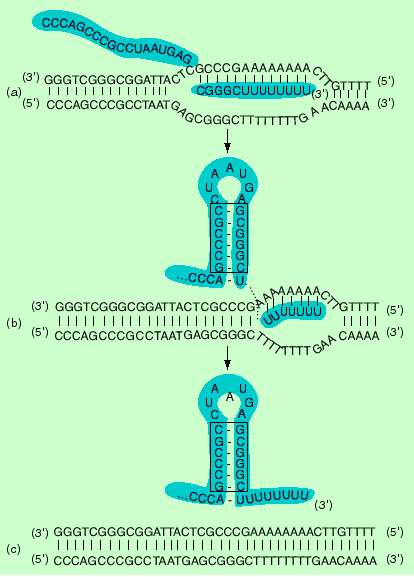
Die RNA-Polymerase bindet schwach an irgendeine Stelle auf der Bakterien-DNA. Von dort gleitet sie der DNA entlang, d.h. sie löst und bindet sich abwechslungsweise, bis sie auf eine Promotor-Sequenz trifft. Dort bleibt sie haften. Bei dieser Bindung spielt die s-Untereinheit der RNA-Polymerase eine wichtige Rolle: sie verringert einerseits die Wechselwirkung der RNA-Polymerase mit unspezifischer DNA und erkennt andererseits (im Verbund des Holoenzyms) die Nukleotid-Sequenzen sowohl in der -10-Region als auch in der -35-Region und vermittelt so die spezifische Bindung der RNA-Polymerase an den Promotor.
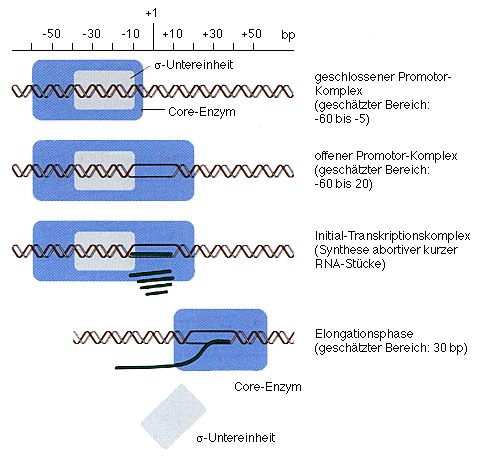
Die Bindung der RNA-Polymerase an den Promotor ist nur der erste Schritt der Initiation. Ihr folgt rasch der Übergang vom geschlossenen in den offenen Promotor-Komplex. Damit einher geht eine Entwindung der DNA Doppelhelix in einem Abschnitt von etwa 12 Basenpaaren um den Transkriptionsstart. Die Konformation der RNA-Polymerase wird verändert, und sie bedeckt nun einen Abschnitt von ungefähr 80 Basenpaaren im Promotor.
Nun beginnt die eigentliche Transkription, indem die RNA-Polymerase ein kurzes Stück RNA synthetisiert, das etwa 10 Nucleotide lang ist. Abhängig von der Art der Promotor-Sequenz und den Bedingungen kann sich eine solche abgebrochene RNA-Synthese vielfach wiederholen, ohne dass die RNA-Polymerase ihre Position verändert oder verlässt: sie steht also still. Man spricht vom Initial-Transkriptionskomplex.
Erst wenn die RNA-Stücke eine Länge von 12 oder mehr Nukleotiden erreichen, geht der Intial-Transkriptionskomplex in den Elongationskomplex über. Die s-Untereinheit fällt ab, das Core-Protein verlässt den Promotor und begibt sich auf den Weg entlang des Matrizenstranges. Die RNA-Polymerase nimmt dabei eine neue Konformation an, denn sie bedeckt nur noch einen DNA Abschnitt von etwa 30 Basenpaaren.
(Abbildung
X
Initiation:Erst wenn die RNA-Stücke Längen von 12 oder mehr Nukleotiden erreichen, geht der Intial-Transkriptionskomplex in den Elongationskomplex über. Die s-Untereinheit fällt ab, das Core-Protein verlässt den Promotor und begibt sich auf den Weg entlang des Matrizenstranges. Die RNA-Polymerase nimmt dabei eine neue Konformation an, denn sie bedeckt nur noch einen DNA Abschnitt von etwa 30 Basenpaaren.
 Bildquelle: Knippers R.: "Molekulare Genetik", 6. neubearbeitete Auflage 1996,
Bildquelle: Knippers R.: "Molekulare Genetik", 6. neubearbeitete Auflage 1996,Georg Thieme Verlag Stuttgart - New York, ISBN 3-13-477006-7
Elongation
Das Prinzip der Kettenverlängerung liegt in der ständigen Wiederholung der Reaktionsfolge: Auswahl des passenden Nukleotids, Anheftung an das 3'-OH-Ende der letzten Base und Bewegung des Enzyms relativ zur DNA-Matrize. Dies läuft bei optimalen Bedingungen mit einer Geschwindigkeit von durchschnittlich 30-60 Nucleotiden pro Sekunde ab (Abbildung
X
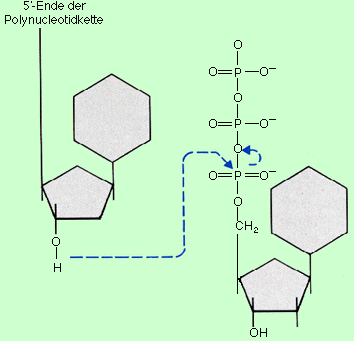
InitiationAlle Nukleinsäuren werden aus Nukleosidtriphosphaten synthetisiert. Die untenstehende Abbildung zeigt die Kondensationsreaktion zwischen der 5' Triphosphatgruppe des hinzukomrnenden Nukleotids und der 3'OH-Gruppe des letzten an die Kette gehängten Nukleotids. Das neue Nukleotid verliert seine zwei endständigen Phosphatgruppen (g und b); seine a-Phosphatgruppe wird für die Phosphodiesterbindung verwendet:
 Bildquelle: Lewin B.:"Gene" 2. Auflage 1991, VCH Verlagsgesellschaft mBH,
Bildquelle: Lewin B.:"Gene" 2. Auflage 1991, VCH Verlagsgesellschaft mBH,Weinheim (D), ISBN 3-527-28052-9
Die Wanderung der RNA-Polymerase entlang der DNA ist keine gleichförmige Bewegung: abhängig von der Nukleotid-Sequenz kann es manchmal zu Verzögerungen oder gar zum Stillstand kommen. Um diese Probleme zu bewältigen, benötigt die RNA-Polymerase zusätzliche Elongationsfaktoren, die mit der aktiven RNA Polymerase in Wechselwirkung treten und sie, je nach Bedarf, wieder verlassen.
Verhalten
X
Verhalten der RNA-Polymerase bei einem Stillstand:Stillstände in ihrer Wanderung bewältigt die RNA-Polymerase durch neue Anläufe; ein endständiges Stück bis zu einer Länge von 9 Nukleotiden wird vom 3'-OH-Ende der RNA abgeschnitten. Dann werden, als neuer Anlauf zur Überwindung der Blockierung, einige neue Nukleotide synthetisiert. Dazu braucht die RNA-Polymerase die Hilfsproteine Gre A und Gre B, die sich bei einem Stillstand an die RNA-Polymerase binden. Weder die RNA-Polymerase noch die Hilfsproteine können das Abschneiden des RNA-Endes allein durchführen. Eine Funktion der Proteine Gre A und Gre B ist die Aktvierung einer Schneideaktivitat der RNA-Polymerase. Die Abtrennung von RNA-Stücken, die bis zu 9 Nukleotide lang sind, setzt eine enge Bindung des wachsenden RNA-Endes mit der RNA-Polymerase voraus. Entsprechende Experimente zeigen, dass die isolierte RNA-Polymerase auch in Abwesenheit von DNA effzient an RNA binden kann.
Termination
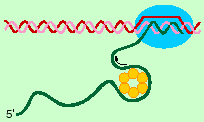
Das Ende eines transkribierten Abschnittes auf der DNA bezeichnet man als Terminator-Region. An dieser Stelle kommt die RNA-Polymerase gezielt zum Stillstand. Es folgt eine Ablösung von RNA und Enzym von der DNA. Bei E. coli unterscheidet man zwei Arten der Termination:
- Rho-abhängige Termination. Das Rho-Protein besteht aus sechs identischen Untereinheiten mit einem Molekulargewicht von je etwa 50 kDa. Es bindet an die gerade gebildete RNA in der Nähe des Terminators und vermittelt so die Ablösung der RNA vom Enzym und von der DNA. Dazu ist die Bereitstellung chemischer Energie in Form von ATP notwendig. Eine der Funktionen des Rho-Proteins ist die Trennung der Wasserstoffbrücken zwischen komplementären DNA- und RNA-Strängen (Helikase-Aktivität).
- Einfache (oder Rho-unabhängige) Termination, wird durch spezifische Sequenzen in der DNA (und damit in der RNA) bestimmt. Eine solche Sequenz findet sich am Ende vieler Transkriptionsabschnitte im Bakteriengenom. Sie besteht aus Folgen von GC-Nukleotiden mit einem anschliessenden Block von Adenin-Resten. Nach der Transkription dieser Terminationssequenz kann die gerade synthetisierte RNA eine Doppelstrangstruktur mit einem Stamm aus 4-10 GC-Basenpaaren und mit einer Schleife von 3-8 Nukleotiden bilden. Vermutlich interagiert der RNA-Doppelstrang mit der RNA-Polymerase und verändert dadurch die Konformation des Enzyms, so dass der Komplex aus RNA, Enzym und DNA auseinanderfällt.
 KnippersR.:
KnippersR.:"Molekulare Genetik",
6. neubearbeitete Auflage 1996,
Georg Thieme Verlag Stuttgart - New York,
ISBN 3-13-477006-7
Schematische Darstellung
X
TerminationEinfache (oder Rho-unabhängige) Termination
 Bildquelle: Lehninger: "Prinzipien der Biochemie"
Bildquelle: Lehninger: "Prinzipien der Biochemie"2. Auflage, Spektrum Akademischer Verlag,
ISBN 3-86025-106-6
b) Durch die intramolekulare Paarung komplementeärer Sequenzen in der RNA wird eine Haarnadel gebildet, wodurch ein Teil des RNA-DNA-Hybrids zerstört wird.
c) Der verbleibende AU-Hybridbereich ist relativ instabil, und die RNA dissoziert vollständig von der DNA ab.
Die Produkte
Alle Klassen von RNA, also mRNA (dient als Matrize bei der Translation), rRNA (ist am Aufbau der Ribosomen beteiligt) und tRNA (sie bindet und liefert bei der Translation die Aminosäuren), werden in E. coli Zellen mit etwa gleicher Rate synthetisiert. Trotzdem ist nur ein Anteil von 5-10 % der Gesamt-RNA einer Bakterienzelle vom Typ der mRNA, 75-80% ist rRNA, der Rest tRNA. Diese Werte mögen erstaunen, wenn man bedenkt, dass zu einer gegebenen Zeit 1000 oder mehr proteinkodierende E. coli-Gene transkribiert werden und mRNA liefern, während nur 7 rRNA-Gene und etwa 50 tRNA-Gene transkribiert werden. Die Ursache liegt darin, dass mRNA bald nach ihrer Synthese wieder abgebaut wird, während rRNA und tRNA für längere Zeit erhalten bleiben.
Die Halbwertszeit durchschnittlicher bakterieller mRNA liegt im Bereich von wenigen Minuten. Dies mag verschwenderisch erscheinen. Es hat aber für Bakterien den Vorteil der grösseren Flexibilität, denn die Bakterienzellen können ihre Genexpression rasch umstellen. Neue Transkripte können produziert werden und in der Zelle zu wirken beginnen, ohne dass ihr Effekt für längere Zeit von der Aktivität früher synthetisierter mRNA überdeckt wird.
Die stabilen RNA-Arten sind Bestandteile des Proteinsynthese-Apparates. Mehr dazu finden Sie im Kapitel "Translation".
Regulation der Transkription
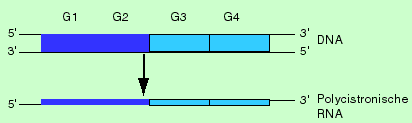
Im Stoffwechsel von Bakterien sind sehr oft mehrere Enzyme an einer biochemischen Reaktionsfolge beteiligt. Ihre Synthese wird erniedrigt, wenn das Substrat fehlt und erhöht, sobald das Substrat erscheint. Aus dieser Verhaltensweise erklären sich die zentralen Merkmale der Organisation bakterieller Gene. Gewöhnlich sind die Gene in Gruppen angeordnet, sog. Operons, welche für mehrere Proteine, die für einen bestimmten Stoffwechselweg benötigt werden, kodieren.
Eine solche Transkriptionseinheit wird in eine einzige polycistronische mRNA transkribiert, die dann durch die Ribosomen in die einzelnen Proteine translatiert wird.
Die Synthese von Enzymen als Antwort auf das Erscheinen eines spezifischen Substrats nennt man Induktion. Dieser Regulationstyp ist bei Bakterien weit verbreitet. Das Musterbeispiel für diese Art von Kontrollmechanismen liefert das Lactose-Operon von E. coli.
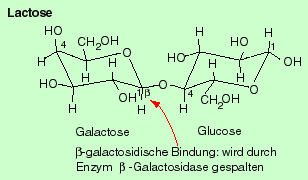
Wenn man E. coli Zellen in Abwesenheit eines b-Galaktosids wachsen lässt, enthalten sie sehr wenige Moleküle des Enzyms b -Galaktosidase - vermutlich weniger als 5. Die Aufgabe dieses Enzyms ist es, b -Galaktoside in seine Zuckerkomponenten zu spalten; z.B. wird Lactose in Glucose und Galactose gespalten, welche dann weiter abgebaut werden. In Abwesenheit des Substrates ist kein Bedarf für das Enzym.
Lactose-Spaltung:
KnippersR.:
"Molekulare Genetik",
6. neubearbeitete Auflage 1996,
Georg Thieme Verlag Stuttgart - New York,
ISBN 3-13-477006-7
Das Lac-Operon von E. coli: Beispiel einer Enzyminduktion
Das Lac-Operons enthält die drei Strukturgene lacZ, lacY und lacA. Es sind die Gene, die die Information zur Synthese von Enzymen des Lactose-Stoffwechsels tragen. Die Aktivität dieser Gene unterliegt einer genauen Regulation, denn im lactose-freien Nährmedium werden in einer Bakterienzelle nur wenige Moleküle der Lactose verwertenden Enzyme gebildet. In Gegenwart von Lactose als einziger Kohlenstoffquelle nimmt die Menge an Lactose-Enzymen um mehr als das 1000-fache zu. Der Grund dafür ist, dass die von der Zelle aufgenommene Lactose bzw. ein Umwandlungsprodukt (Allolactose) die Transkription des Lac-Operons anregt.
Die drei Gene des Lac-Operons:
- lacZ kodiert für das Enzym b -Galactosidase, dessen aktive Form ein Tetramer aus 4 Untereinheiten von gesamthaft ca. 500'000 Dalton ist. Jede Untereinheit hat 1023 Aminosäuren. Damit ist die b -Galactosidase eines der grössten Proteine der Bakterienzelle. Sie spaltet das Disaccharid Lactose in Glucose und Galactose, die dann jeweils über eigene Stoffwechselwege zur Gewinnung zellulärer Energie abgebaut werden.
- lacY codiert für die b -Galactosid-Permease, eine membrangebundene 30'000 Dalton schwere Proteinkomponente des Transportsystems. Dieses Carrier-Protein erleichtert der Zelle die Aufnahme von Lactose.
- lacA codiert für die b -Galactosid-Transacetylase, ein Enzym, das eine Acetylgruppe von Acetyl-CoA auf b -Galactoside überträgt. Die physiologische Bedeutung dieses Enzyms für den Bakterienstoffwechsel ist nicht bekannt.
Mutationen
X
Das lac-Operon von E. coli: Beispiel einer EnzyminduktionMutationen:
Mutationen sowohl im lacZ als auch im lacY können den lac~ -Genotyp erzeugen, bei dem die Zellen keine Lactose verwerten können. Die lacZ~ Mutationen heben die Enzymaktivität auf und verhindem so direkt den Metabolismus von Lactose. Die lacY~ -Mutanten können Lactose nicht aus dem Medium aufnehmen. Bei lacA~ -Zellen ist kein Defekt zu erkennen.
Ausser Lactose können mehrere andere Galactoside die Synthese der drei Proteine induzieren, darunter sind auch solche Galactoside, die von der b-Galactosidase nicht gespalten werden können. Der stärkste Induktor, Isopropyl-b-D-thiogalactosid (IPTG), gehört zu dieser Gruppe von Galactosiden.
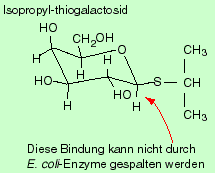
Der Aufbau des Lactose-Operons:
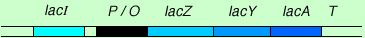
P / O: Prornotor-Operator-Region
T: Terminator
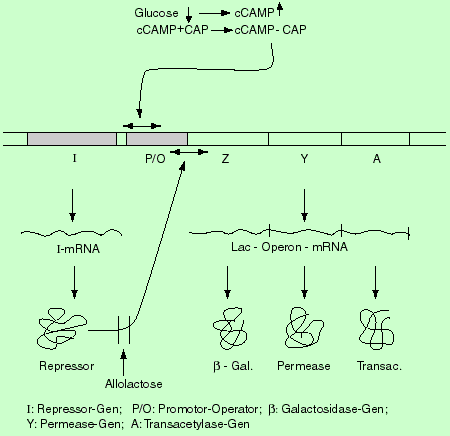
Die Regulation der Transkription des Lactose-Operons
Die drei Gene lacZ, Y, und A werden in eine einzige mRNA transkribiert, ausgehend vom Promotor Plac. Die Möglichkeit, die Transkription bei Plac zu initiieren, wird durch das Repressorprotein kontrolliert, welches vom Regulatorgen lacI kodiert wird.
Bildquelle: Prof. Hans Trachsel: "Einführung in die Molekularbiologie -
Skript für Studierende der Human- und Veterinärmedizin",
Institut für Biochemie und Molekularbiologie der Universität Bern, 1996
Der Repressor bindet an den Operator und verhindert damit die Transkription des Operons. Der Operator befindet sich zwischen Promotor und dem Leseraster für lacZ. Die Bindung des Repressors an diese Stelle hindert die RNA-Polymerase an der Initiation der Transkription am Promotor.Gibt man einen Induktor hinzu, so startet die Transkription an Plac und läuft über die Gene bis zum Terminator hinter lacA. Damit ergibt sich koordinierte Regulation: alle Gene werden miteinander exprimiert (oder nicht exprimiert). Die lac-mRNA ist instabil und wird mit einer Halbwertszeit von ca. drei Minuten abgebaut. Diese Eigenschaft erlaubt eine schnelle Umkehrung der Induktion: die Transkription hört auf, sobald der Induktor entfernt wird; in sehr kurzer Zeit ist sämtliche lac-mRNA zerstört, und die Zelle stoppt die Produktion der Enzyme.
Das Lac-Operon enthält das Strukturgen lacZ für die b-Galactosidase und das Gen lacY für das Transportprotein (Permease), welches das Substrat in die Zelle führt. Wie soll jedoch der Induktor in die Zelle gelangen, um den Prozess der Induktion zu starten, wenn sich das Operon in reprimiertem Zustand befindet? Zwei Prozesse dürften sicherstellen, dass immer minimale Mengen b-Galactosidase und Permease in der Zelle vorhanden sind, welche ausreichen, die Transkription in Gang zu setzen: Die Expression des Operons wird auf einem Grundniveau gehalten; sogar wenn die Transkription nicht induziert ist, wird das Operon mit einer Minimalrate exprimiert (ca. 0.1% des induzierten Niveaus). Ausserdem können einige Induktoren möglicherweise über ein anderes Aufnahmesystem in die Zelle eindringen.
Das Repressorprotein besitzt eine sehr hohe Affinität zum Operator. In Abwesenheit des Induktors bindet es dort. Wenn aber der Induktor vorhanden ist, bindet dieser den Repressor und bildet einen Repressor/lnduktor-Komplex, welcher nicht mehr an den Operator bindet. Somit hat der Repressor eine Doppelfunktion: einerseits kann er die Transkription verhindern, andererseits kann er den Induktor erkennen. Wenn der Induktor bindet, verändert er die Konformation des Repressors derart, dass die Affinität für die Bindungsstelle an der DNA stark herabgesetzt wird.
Neben der oben beschriebenen Repression existiert für das Lac-Operon auch der andere Typ der Regulation, nämlich die Stimulation. Bei der Stimulation wird die Transkription des Operons dramatisch verstärkt. Dies ist der Fall, wenn im Medium Lactose vorhanden ist, aber keine Glucose. Bei Glucose-Mangel steigt die cAMP Konzentration (zyklisches AMP) stark an. cAMP bindet an ein Rezeptor-Protein (CAP=cAMP-bindendes Protein). Dieser Komplex bindet dann an den Operator, stimuliert die Bindung der RNA-Polymerase und sorgt damit für eine verstärkte Transkription.