Mechanismus der Translation
Die Vorgänge bei der Translation werden in drei Phasen aufgeteilt: Initiation, Elongation und Termination.
Die Initiation ist bei Prokaryoten und Eukaryoten unterschiedlich:
Initiation bei E. coli
Die kleine 30S Ribosomenuntereinheit muss sich an das zu translatierende mRNA-Molekül anlagern, und zwar nicht an einer beliebigen Position, sondern an einer spezifischen Stelle unmittelbar stromaufwärts (Richtung 5') vom Translationsstartcodon der mRNA. Der richtige Ort für die Anlagerung ist durch eine Nukleotidsequenz bestimmt, welche in E. coli folgendendermassen lautet:
5'-AGGAGGU-3'
Diese als Shine-Dalgarno-Sequenz (S/D-Sequenz) bekannte Nukleotidfolge geht eine vorübergehende Basenpaarung mit einem Teil des 16S rRNA ein und ermöglicht so der 30S Untereinheit, an die mRNA zu binden.
Da die kleine Untereinheit einen viel grösseren Bereich abdeckt als die Länge der Shine-Dalgarno-Sequenz, ist mit der Identifikation der Ribosomenbindungsstelle auch gleich das Startcodon AUG korrekt positioniert. Das Startcodon liegt nämlich üblicherweise nur etwa 10 Nukleotide hinter der S/D-Sequenz. Das AUG-Triplett ist das Translationsstartcodon der mRNA, d.h. es markiert die Stelle, an der die Translation beginnen muss.
Prokaryoten können mehrere Proteine auf einer einzigen mRNA codieren und somit gleichzeitig verschiedene Proteine von derselben mRNA synthetisieren. Solche polycistronische mRNAs benötigen deshalb auch dementsprechend viele Shine-Dalgarno-Sequenzen (S/D) und ebensoviele Start- und Stop-Codons:
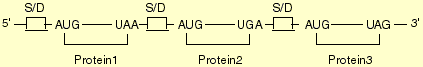
Der Translationsvorgang selbst beginnt, sobald eine Initiator-tRNA mit dem Start-Codon, das durch die 30S Untereinheit ausfindig gemacht wurde, eine Basenpaarung eingeht. Diese Initiator-tRNA ist bei Bakterien mit N-Formylmethionin (fMet) beladen:
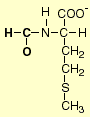
Den Komplex aus mRNA, 30S Untereinheit und Formylmethionin-tRNA nennt man Initiationskomplex, und seine Bildung markiert das Ende der Initiationsphase der Translation.
Synthese von Formylmethionin
X
Synthese von FormylmethioninObwohl es für Methionin nur ein Codon gibt (AUG), besitzen alle Organismen zwei tRNA-Typen zur Bindung von Methionin. Eine tRNA wird ausschliesslich dann verwendet, wenn AUG das Initiations-Codon für die Proteinsynthese dient. Die zweite tRNA wird verwendet, wenn Methionin in eine interne Position eines Polypeptids eingefügt wird.
Bei Bakterien werden die beiden Typen von methionin-spezifischer tRNA als tRNAMet und tRNAfMet bezeichnet. Der erste Aminosäurerest am amino-terminalen Ende ist N-Formylmethionin. Er wird als N-Formyl-methionin-tRNAfMet, das in zwei aufeinanderfolgenden Reaktionen gebildet wird, zum Ribosom transportiert. Zuerst wird Methionin von der Met-tRNA-Synthetase an die tRNAfMet gebunden:
Methionin + tRNAfMet + ATP -> Met- tRNAfMet + AMP + PPi
Darauf wird eine Formylgruppe von N10-Formyltetrahydrofolat durch eine Transformylase auf die Aminogruppe des Methionin-Restes übertragen:
N(10)-Formyltetrahydrofolat + Met- tRNAfMet -> Tetrahydrofolat + fMet- tRNAfMet
Diese Transformylase kann kein freies Methionin oder an tRNAMet gebundenes Methionin formylieren. Statt dessen ist sie spezifisch für an tRNAfMet gebundene Methionin-Reste, wobei sie ein charakteristisches Strukturmerkmal dieser tRNA erkennt.
Die fMet-tRNAfMet wird vom Translationsinitiationsfaktor IF2 erkannt und an die P-Region der 30S ribosomalen Untereinheit gebunden.
Initiation bei Eukaryoten
Der wesentliche Unterschied zu Prokaryoten betrifft die Art, wie die kleine Untereinheit des Ribosoms (40S bei Eukaryoten) an die mRNA bindet und das AUG-Codon, an dem die Translation beginnen soll, ausfindig macht. Ausserdem sind für die Initiation mindestens elf Initiationsfaktoren erforderlich, im Gegensatz zu E. coli, wo drei solche Proteine ausreichen.
Bei Eukaryoten bindet einer dieser Initiationsfaktoren, das cap-bindende Protein eIF4E, an die 5'-Cap-Struktur der mRNA und fördert die Bildung eines Komplexes zwischen der mRNA und der 40S Untereinheit des Ribosoms. Die 40S Untereinheit hat bereits Initiator-Methionyl-tRNA gebunden. Die 40S Untereinheit wandert dann der mRNA entlang, bis sie ein Start-Codon erreicht. Dort wird die 60S Untereinheit des Ribosoms angefügt, und es resultiert ein 80S-Initiationskomplexes, der die Initiator-Met-tRNAMet sowie mRNA enthält und für die Elongation bereit ist.
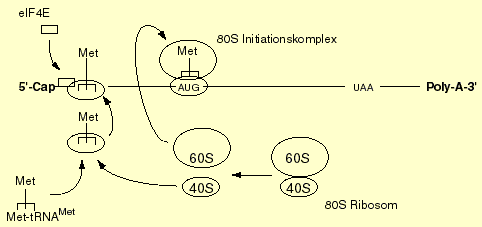
Aufgabe: Beschreiben Sie, wie die 40S Untereinheit eines eukaryotischen Ribosoms ein Startcodon auf einem mRNA-Molekül findet.
Elongation
Die Elongation verläuft bei Prokaryoten und Eukaryoten gleich:
Sobald sich der Initiationskomplex gebildet hat, kann sich die grosse Untereinheit des Ribosoms anlagern. Es entstehen so zwei verschiedene abgegrenzte Bereiche, an denen tRNAs binden können: Die erste heisst Peptidyl- oder P-Stelle und ist im Augenblick von der Methionin-tRNA (bei Prokaryoten: Formyl-methionin-tRNA) besetzt, die noch mit dem Start-Codon basengepaart ist. Die Aminoacyl- oder A-Stelle liegt über dem zweiten Codon des Gens und ist zunächst unbesetzt.
Den Prozess der Polypeptidketten-Verlängerung (Elongation) kann man sich als zyklischen Vorgang mit drei klar voneinander unterscheidbaren Schritten betrachten:
- Bindung einer neuen tRNA in Position A
Eine Aminoacyl-tRNA plus GTP wird an den Elongationsfaktor eEF1 (EF-Tu in E. coli) gebunden und dieser Komplex findet die leere A-Stelle neben einer besetzten P-Stelle am Ribosom. Dort bildet das Anticodon der tRNA Basenpaare mit drei Nukleotiden (Codon) der mRNA. Das GTP-Molekül wird hydrolysiert und der Elongationsfaktor entlassen. - Bildung der Peptidbindung
Im zweiten Schritt wird das Carboxy-Ende der Polypeptidkette von dem an der P-Bindungsstelle liegenden tRNA-Molekül getrennt und über eine Peptidbindung an die Aminosäure gebunden, die an das tRNA-Molekül in der A-Stelle gebunden ist. Katalysiert wird diese Reaktion von der Peptidyltransferase. Diese enzymatische Aktivität wird durch eine Base der 28S rRNA in der grossen Ribosomen-Untereinheit vermittelt. - Translokation und Freisetzung der tRNA
Im dritten Schritt wird schliesslich die neue Peptidyl-tRNA von der A-Stelle in die P-Stelle verschoben. Dieser Schritt wird durch den Elongationsfaktor eEF2 (EF-G in E. coli) und GTP katalysiert. Dabei bewegt sich das Ribosom um genau drei Nukleotide auf der mRNA weiter und das das GTP-Molekül wird hydrolysiert. Während des Translokationsvorgangs löst sich das im zweiten Schritt an der P-Stelle gebildete freie tRNA-Molekül vom Ribosom und kehrt in den cytoplasmatischen tRNA-Vorrat zurück. Daher ist am Ende des dritten Schritts die A-Stelle wieder frei und kann eine neue Aminoacyl-tRNA aufnehmen. Damit beginnt der ganze Vorgang von neuem.
Neu: Peptidyltransferase
X
PeptidyltransferaseEnzymatische Aktivität
- Katalysiert die Bildung der neuen Peptidbindung
- Ist auf der grossen ribosomalen Untereinheit lokalisiert
Die Peptidyltransferase ist ein Ribozym
- Eine Adeninbase der ribosomalen RNA katalysiert die Bildung der Peptidbindung
Bedeutung für unsere Vorstellungen über die Evolution
- RNA katalysiert heute noch die Synthese von Eiweissen
- Kleine RNA-Fragmente waren wahrscheinlich an der Synthese der ersten Eiweisse beteiligt
Schema
X
Schema zur Elongation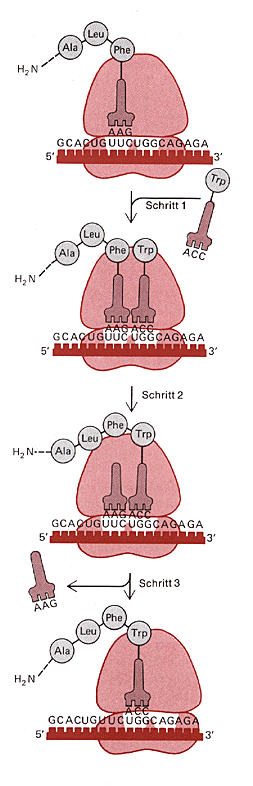
- Bindung einer neuen tRNA in Position A
Im ersten Schritt wird ein Aminoacyl-tRNA-Molekül an einer leeren A-Stelle neben einer besetzten P-Stelle an das Ribosom gebunden. Dort bildet es Basenpaare mit den drei mRNA-Nukleotiden, die an der A-Stelle exponiert sind. - Bildung der Peptidbindung
Im zweiten Schritt wird das Carboxy-Ende der Polypeptidkette von dem an der P-Bindungsstelle liegenden tRNA-Molekül getrennt und über eine Peptidbindung an die Aminosäure gebunden, die an das tRNA-Molekül in der A-Stelle gebunden ist. Katalysiert wird diese Reaktion von der Peptidyltransferase. Diese enzymatische Aktivität wird vermutlich durch einen bestimmten Abschnitt des grössten rRNA-Moleküls in der grossen Ribosomen-Untereinheit vermittelt. - Translokation und Freisetzung der tRNA aus Position B
Im dritten Schritt schliesslich wird die neue Peptidyl-tRNA von der A-Stelle in die P-Stelle verschoben. Dabei bewegt sich das Ribosom um genau drei Nukleotide auf der mRNA weiter. Dieser Schritt erfordert Energie und wird durch eine Folge von Konformationsänderungen in Gang gesetzt, die durch Hydrolyse eines GTP-Moleküls getrieben werden. Während des im dritten Schritt ablaufenden Translokationsvorgangs löst sich das im zweiten Schritt an der P-Stelle gebildete freie tRNA-Molekül vom Ribosom und kehrt in den cytoplasmatischen tRNA-Vorrat zurück. Daher ist am Ende des dritten Schritts die A-Stelle wieder frei und kann eine neue tRNA aufnehmen, an welche die nächste Aminosäure gebunden ist. Damit beginnt der ganze Vorgang von neuem.
Termination
Auch die Termination ist bei Prokaryoten und Eukaryoten im Wesentlichen gleich:
Sobald ein Stopcodon (UAA, UAG oder UGA) in die A-Stelle gelangt, kommt es zur Termination der Translation. Es gibt kein tRNA-Molekül, dessen Anticodon mit einem der Stop-Codons eine Basenpaarung eingehen könnte. Stattdessen besetzt einer von zwei Terminationsfaktoren (eRF1 oder eRF2, bei Eukaryoten) die A-Stelle des Ribosoms. Dabei erkennt eRF1 nur die Stop-Codons UAG und UAA, eRF2 dafür UGA und UAA. Ein dritter Terminationsfaktor (eRF3) erfüllt bei diesem Vorgang eine Hilfsfunktion. Die Bindung von eRF an die A-Stelle ändert die Aktivität der Peptidyltransferase so, dass diese ein Wassermolekül anstelle einer Aminosäure an die Peptidyl-tRNA anhängt. Dadurch wird das Carboxy-Ende der Polypeptidkette aus der Bindung an das tRNA-Molekül gelöst. Da normalerweise die wachsende Polypeptidkette ausschliesslich durch diese Bindung mit dem Ribosom verknüpt ist, wird die fertige Proteinkette ins Cytoplasma entlassen. Anschliessend setzt das Ribosom auch die mRNA und die tRNA der zuletzt eingebauten Aminosäure frei und zerfällt in seine beiden Untereinheiten.Diese können sich sogleich wieder an eine mRNA anlagern.
Schema
X
Schema zur Elongation
- Bindung einer neuen tRNA in Position A
Im ersten Schritt wird ein Aminoacyl-tRNA-Molekül an einer leeren A-Stelle neben einer besetzten P-Stelle an das Ribosom gebunden. Dort bildet es Basenpaare mit den drei mRNA-Nukleotiden, die an der A-Stelle exponiert sind. - Bildung der Peptidbindung
Im zweiten Schritt wird das Carboxy-Ende der Polypeptidkette von dem an der P-Bindungsstelle liegenden tRNA-Molekül getrennt und über eine Peptidbindung an die Aminosäure gebunden, die an das tRNA-Molekül in der A-Stelle gebunden ist. Katalysiert wird diese Reaktion von der Peptidyltransferase. Diese enzymatische Aktivität wird vermutlich durch einen bestimmten Abschnitt des grössten rRNA-Moleküls in der grossen Ribosomen-Untereinheit vermittelt. - Translokation und Freisetzung der tRNA aus Position B
Im dritten Schritt schliesslich wird die neue Peptidyl-tRNA von der A-Stelle in die P-Stelle verschoben. Dabei bewegt sich das Ribosom um genau drei Nukleotide auf der mRNA weiter. Dieser Schritt erfordert Energie und wird durch eine Folge von Konformationsänderungen in Gang gesetzt, die durch Hydrolyse eines GTP-Moleküls getrieben werden. Während des im dritten Schritt ablaufenden Translokationsvorgangs löst sich das im zweiten Schritt an der P-Stelle gebildete freie tRNA-Molekül vom Ribosom und kehrt in den cytoplasmatischen tRNA-Vorrat zurück. Daher ist am Ende des dritten Schritts die A-Stelle wieder frei und kann eine neue tRNA aufnehmen, an welche die nächste Aminosäure gebunden ist. Damit beginnt der ganze Vorgang von neuem.
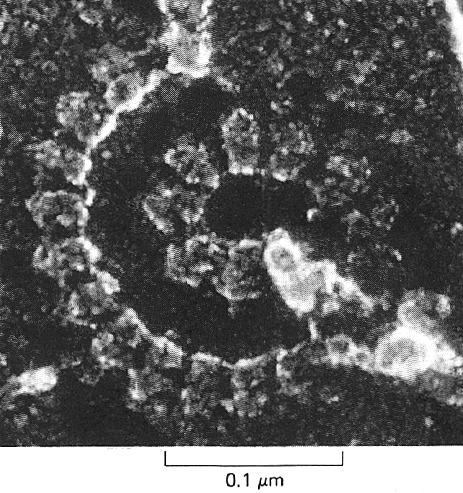
Ein mRNA ist so gross, dass mehrere Ribosomen sie gleichzeitig übersetzen können. Solche Ribosomen-mRNA-Komplexe nennt man Polyribosomen oder auch Polysomen. Sie ermöglichen eine rasche und intensive Proteinsynthese. Polysomen können elektronenmikroskopisch sichtbar gemacht werden: