Die Transkription bei Eukaryonten
Die Transkription bei Eukaryonten verläuft nach den gleichen Prinzipien wie die Transkription bei den Prokaryoten; es gibt jedoch einige wichtige Unterschiede:
Chromatin-Remodellierung
Die DNA eukaryotischer Zellen ist als Chromatin organisiert. Die Transkription einzelner Gene bedingt vorgängige Veränderungen in der lokalen Chromatinstruktur, welche Gene für den Transkriptionsapparat zugänglich machen:
- Jede Zelle transkribiert zu einem gewissen Zeitpunkt nur einen Teil ihres
Genoms - Nicht-transkribierte Gene und benachbarte DNA-Regionen sind sehr dicht gepackte DNA-Protein-Komplexe (Heterochromatin)
- Transkribierte Gene und benachbarte DNA-Regionen sind weniger dicht gepackt (Euchromatin) und teilweise sogar frei von Histonen
- Die Aufhebung der Heterochromatin-Struktur wird durch DNA-bindende Proteine und Acetylierung von Histonen erreicht. Diese Vorgänge werden als Chromatin-Remodellierung bezeichnet und sind Voraussetzung für die Transkription
RNA Polymerasen
Im Gegensatz zu prokaryotischen Zellen besitzen eukaryotische Zellen nicht nur eine, sondern gleich drei verschiedene RNA-Polymerasen: RNA-Polymerase I, II und III.
- Die RNA-Polymerase I befindet sich im Nucleolus und transkribiert diejenigen Gene, die für ribosomale RNA (18S, 28S) kodieren.
- Die RNA-Polymerase II transkribiert vorwiegend Gene, welche für Proteine kodieren.
- Die RNA-Polymerase III synthetisiert die kleineren RNAs wie tRNA, ribosomale 5S-RNA und einen Teil der kleinen Kern-RNA-Arten (snRNA=small nuclear RNA).
Wir wollen nur die RNA-Polymerase II-Gene näher betrachten.
Eukaryotisches protein-kodierendes Gen
Das Genom des Menschen enthält schätzungsweise 30'000-50'000 Gene, welche für Eiweisse kodieren. In ihrem grundsätzlichen Aufbau gleichen sie den prokaryotischen Genen:
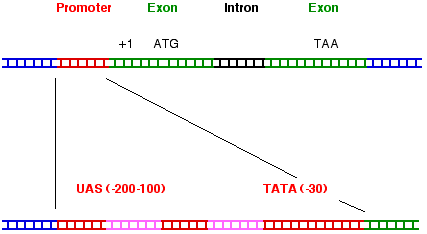
Transkribierte Region: Der Anfang der transkribierten Region, Position +1, heisst cap site, weil nach dem Beginn der Transkription die entstehende mRNA am ersten Nukleotid durch Anhängen der Cap-Struktur modifziert wird. Die Cap-Struktur wird durch Uebertragung von GTP auf die 5' terminale Base und Methylierung von Guanin in Position 7 synthetisiert. Die Cap-Struktur schützt die mRNA gegen Abbau durch Nukleasen und ist wichtig für die Translation.
Termination: Nach dem Passieren der Poly(A)-Addierungs-Sequenz AAUAAA auf der neusynthetisierten RNA pausiert die RNA-Polymerase II, assoziierte Proteine erkennen die Sequenz AAUAAA und die Transkription stoppt. Ein Enzymkomplex bindet an die Poly(A)-Addierungs-Sequenz AAUAAA der RNA, schneidet sie 10 - 15 Nukleotide weiter hinten (3' -Richtung) und hängt 50-150 Adenosine (Poly(A)-Sequenz) an.
Anordnung von Genen: In höheren Eukaryoten finden wir sehr oft Gene, die für das gleiche oder sehr ähnliche Proteine kodieren, sog. Gen-Familien. In der Regel werden nur wenige Gene, oft gar nur ein Gen aus einer Gen-Familie exprimiert. Die nicht-funktionellen Mitglieder der Gen-Familie werden als Pseudogene bezeichnet. Eukaryoten haben, im Gegensatz zu Prokaryoten, Gene nicht in Operons organisiert.
Genom-Grössen: Vergleicht man die Genom-Grössen von Zellen, so stellt man fest, dass sie mit zunehmendem Entwicklungsstand während der Evolution zunehmen. Eigenartigerweise stimmt diese Beziehung innerhalb der Eukaryoten nicht generell. So variiert die Genom-Grösse innerhalb der Amphibien von 109 bis 1011 Basenpaaren. Zudem sind die Genome grösser, als man aus der Anzahl Gene (10'000 - 100'000) in eukaryotischen Zellen erwarten würde. Aus dieser Beobachtung ergibt sich, dass nur ein kleiner Teil des Genoms von höheren Eukaryoten für Protein kodiert. Was ist die restliche DNA und welche Funktion hat sie? Die Genome von höheren Eukaryoten enthalten neben den einzelnen Genen noch hoch- und mittel-repetierte DNA-Sequenzen. Zu den hochrepetierten Sequenzen gehören DNA-Abschnitte von 300 - 1000 Basenpaaren Länge, die bis zu 100'000-fach wiederholt vorkommen. Ihre Funktion ist nicht bekannt. Zu den mittel-repetierten Sequenzen gehören die Gene für ribosomale RNA und tRNAs. Diese Gene sind repetiert, weil grosse Mengen der entsprechenden Gen-Produkte von der Zelle benötigt werden.
NEU
X
Kurze regulatorische RNAsKurze regulatorische RNAs wurden erst Ende des 20. Jahrhunderts entdeckt. Sie sind 21-23 Nukleotide lang und regulieren die Genexpression auf verschiedenen Stufen:
- sie bewirken die Bindung von Eiweissen an bestimmte DNA-Regionen und via Modifikation von Histonen die Veränderung der Chromatin-Struktur und der Genexpression auf Stufe Transkription.
- sie hybridisieren an mRNA und regulieren deren Lebensdauer und/oder Uebersetzung in Protein (Translation).
Biogenese von kurzer regulatorische RNA:
- Neben Genen, welche für Proteine oder rRNA und tRNA kodieren, gibt es noch Gene, welche für sog. "microRNA" (miRNA) kodieren. Von diesen Genen werden Vorläufer-RNAs abgelesen, welche auf sich selber Doppelstrangstrukturen bilden können. Von diesen RNA-Doppelsträngen wird durch einen Proteinkomplex (welcher eine RNase namens DICER enthält) eine 21-23 Nukleotidpaare lange Doppelstrang-RNA abgespalten und aus dieser miRNA freigesetzt (wahrscheinlich durch RISC, siehe unten). Beispiel:
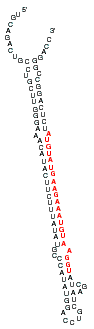
Fig.: Vorläufer für eine humane miRNA. Der rote Anteil ist die reife miRNA.
- Transcripte von gewissen DNA-Regionen werden durch eine RNA-abhängige RNA-Polymerase in doppelsträngige RNA überführt, von DICER in 21-23 Nukleotidpaare lange doppelsträngige RNAs gespalten (siRNA, small interfering RNA) und diese vom Proteinkomplex RISC (RNA-induced silencing complex) in einzelsträngige kurze regulatorische RNAs überführt.
Fragiles X-Syndrom und kurze regulatorische RNA:
- Das Fragile X-Syndrom ist eine Erbkrankheit, die nur bei Knaben und Männern phänotypisch in Erscheinung tritt (schwere Störungen der neuronalen Entwicklung und andere Symptome).
- Diese Erbkrankheit beruht auf der Veränderung eines Gens auf dem X-Chromosom. Das Gen kodiert für das Eiweiss FMR1.
- 2002 entdeckt: In der Fruchtfliege (Drosophila) ist FMR1 Teil des Proteinkomplexes RISC! Dieser Befund impliziert (1) dass das humane FMR1 ebenfalls mit RISC assoziiert sein dürfte und (2) dassVeränderungen in der Bildung von kurzen regulatorischen RNAs zu schweren physiologischen Störungen beim Menschen führen können.
Regulation der Transkription
Die Regulation der Transkription ist aus mehreren Gründen von grosser Bedeutung:
- Der gesamte Prozess der Transkription und Translation ist sehr energieaufwendig und sollte daher auf das notwendige Mass beschränkt sein.
- Durch verstärkte oder verminderte Synthese eines Enzyms kann dessen Konzentration in der Zelle erhöht resp. erniedrigt und dadurch der Stoffwechsel reguliert werden.
- Bei höheren Organismen sind viele Merkmale, die im Verlaufe der Entwicklung ausgebildet werden, genetisch determiniert. Die hierfür verantwortlichen Gene müssen also in bestimmten Entwicklungsphasen "angeschaltet" werden, um ihre Funktion zu erfüllen.
Die Genregulation bei Eukaryoten wurde erst durch die Methoden der Analyse, Sequenzierung und Veränderung (Deletion, Mutation) definierter Genabschnitte einer gezielten Untersuchung zugänglich. Ein Ansatz bestand zunächst darin, DNA Strukturen in der Umgebung gleichartig regulierter Gene miteinander zu vergleichen und strukturell ähnliche Abschnitte zu benennen und ihre Funktion zu analysieren.
Durch Verwendung der geschilderten Methoden ist es gelungen, DNA-Abschnitte zu beschreiben, die die Expression eines Gens beeinflussen. Die TATA-Box und UAS als Teil der Promotorstrukturen wurde bereits genannt. Eine Sequenzklasse, die zunächst bei Eukaryonten-Viren beschrieben, dann aber auch für zelluläre Gene gefunden wurde, sind die Enhancer-Elemente, welche die Aktivität der Promotoren drastisch steigern.
Wichtige Tatbestände zur Genregulation:
- Transkriptionsfaktoren regulieren die Bindung der RNA-Polymerase an Promotoren.
- Die allgemeinen Transkriptionsfaktoren sind für die Ablesung aller Gene notwendig: Der Faktor IID bindet an die TATA-Box von Polymerase II-Genen und ermöglicht damit die Bindung weiterer Faktoren (TFI, IIA, 2B, etc.) in einem grossen Protein-Komplex. Dieser Transkriptionsfaktor-Komplex ist nötig, aber nicht ausreichend, für effiziente Erkennung des Promoters durch die RNA-Polymerase. Es braucht noch spezifische Transkriptionsfaktoren, welche eine DNA-Bindungsstelle haben, mit welcher sie die UAS (upstream activating sequences) erkennen, und eine Sequenz, mit welcher sie die allgemeinen Faktoren binden. Es ist dann dieser Superkomplex aus allgemeinen und spezifischen Faktoren, welcher die RNA-Polymerase bindet und damit die Transkription des Gens ermöglicht:
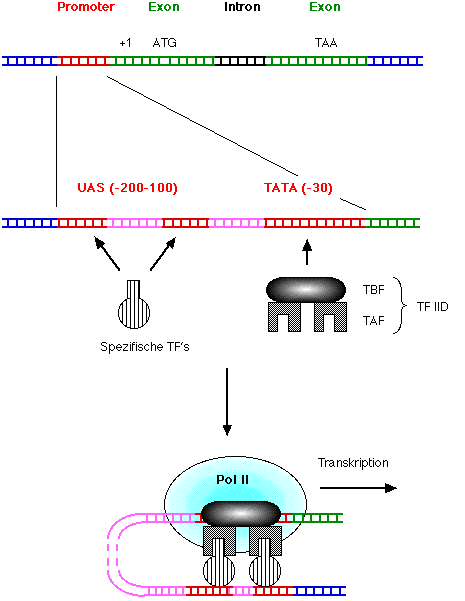
Fig.: TF, Transkriptionsfaktor; UAS, upstream activating sequence; TBF, TATA-binding factor; TAF, TATA-associated factor; Pol II, RNA-Polymerase II.
- Unter den spezifischen Faktoren gibt es neben den stimulierenden auch inhibierende Faktoren. Es ist dann für jedes Gen die Kombination der stimulierenden und inhibierenden Faktoren, welche den Grad der Ablesung bestimmen
- Da die Anwesenheit und die Aktivität der spezifischen Transkriptionsfaktoren durch die Zelle verändert werden kann, ergibt sich für jedes Gen die Möglichkeit der vielfältigen Regulation. Diese Regulation kann zudem durch andere Zellen im Organismus beeinflusst werden. So können z.B. Wachstumsfaktoren oder Hormone an Rezeptoren der Zelle binden und über eine Kaskade von Proteinen die Aktivität von Transkriptionsfaktoren beeinflussen. Sehr oft liegen spezifische Transkriptionsfaktoren in inaktiver Form im Zytoplasma der Zelle und die Aktivierung führt zum Transport des Faktors in den Zellkern:
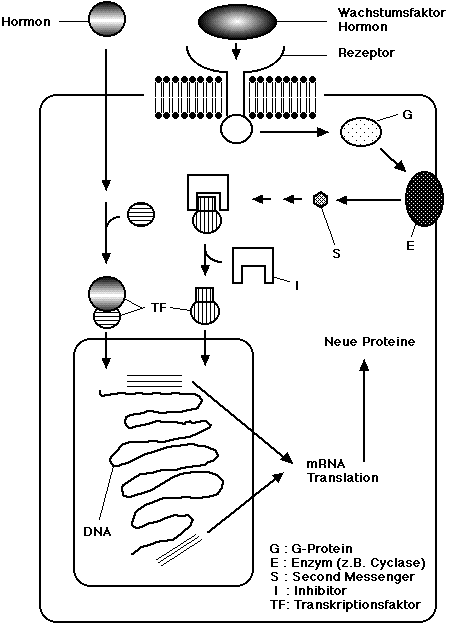
Figur: Die Bindung des Hormons an den Rezeptor führt zur Aktivierung einer Signaltransduktionskette.
- Transkription findet wahrscheinlich an bestimmten Stellen im Zellkern statt: transkribierte und nicht-transkribierte DNA-Abschnitte sind räumlich getrennt.
- Der Transkriptionsort im Kern für DNA,
welche für rRNA kodiert, ist der Nukleolus
Introns, Exons und Splicing
Die Vorstufen der meisten eukaryotischen mRNA-Moleküle enthalten die kodierende Sequenz nicht in ununterbrochener Reihenfolge. Es sind vielmehr ein oder mehrere Stücke nicht in Protein zu übersetzende Basensequenzen eingeschoben. Dies bedeutet, dass die Gene, an denen diese Prä-mRNA synthetisiert wurde, durch entsprechende DNA-Abschnitte unterbrochen sind. Man bezeichnet die nicht-kodierenden Sequenzen als Introns und die kodierenden Sequenzen als Exons.
Das primäre Transkript der Polymerase II ist eine hochmolekulare RNA, die auch Prä-mRNA genannt wird. Die Umwandlung (Prozessierung) der Prä-mRNA zur reifen mRNA beginnt damit, dass am 5'-Ende die sog. Cap-Struktur angehängt wird (siehe oben).
Die zunächst transkribierten Intronabschnitte werden im Verlaufe der Reifung der Prä-mRNA herausgeschnitten, und die kodierenden Abschnitte der mRNA werden zusammengefügt. Diesen Vorgang, den man in Analogie zum Spleissen von Tauwerk als das Splicing der RNA bezeichnet.
Wir unterscheiden 3 Typen (I,II und III) von Introns in eukaryotischen Genen. Sie unterscheiden sich vor allem in der Art und Weise, wie sie aus der Vorläufer-RNA entfernt werden. Nur Typ III kommt in protein-kodierenden Genen vor:
- Typ I schneidet sich selber heraus ("self-splicing", katalytische RNA).
- Typ II benötigt Proteine für effizientes Herausschneiden
- Typ III benötigt komplexe Protein-RNA-Partikel (snRNP's, small nuclear ribonucleoprotein particles) zum Splicing. Typ III Introns finden wir in den für Eiweiss kodierenden Genen in höheren Eukaryoten. Die RNA-Sequenzen über die Exon-Intron Grenze am Anfang und am Ende der Introns sind immer ähnlich oder identisch (konserviert). Sie werden durch RNP's erkannt, indem der RNA-Anteil dieser Partikel mit der hnRNA hybridisiert. Die Reaktionen, welche zum Ausschneiden des Introns und Ligieren der Exons führen, sind nicht im Detail bekannt, doch nimmt man heute an, dass diese Reaktionen von den RNA-Anteilen der snRNP's katalysiert werden.
Eine interessante ungeklärte Frage ist diejenige nach der Entstehungsweise von Introns während der Evolution.
Es gibt Prä-mRNAs, welche nicht immer gleich gespleisst werden, d.h. es können von einer hnRNA mehrere unterschiedliche mRNAs angefertigt werden. Dieses Phänomen bezeichnen wir als "Alternatives Spleissen" (alternative splicing). Alternatives Spleissen wird reguliert und kann die kodierende Kapazität eukaryotischer Genome beträchtlich erhöhen.
Fertig modifizierte RNA wird als mRNA (messenger RNA) bezeichnet. Sie wird vom Kern ins Zytoplasma der Zelle transportiert, wo sie von Ribosomen gebunden und transliert wird.