Protein-Targeting bei Eukaryoten
Fast alle Proteine entstehen an den Ribosomen im Cytosol (nur relativ wenige werden an den Ribosomen der Mitochondrien und Chloroplasten gebildet). Der Weg, welchen die Proteine dann nehmen, teilt sich in zwei Richtungen. Auf dem einen Weg (im Schema unten gestrichelt dargestellt) werden die Proteine, wenn sie fertig synthetisiert sind, ins Cytosol entlassen. Manche dieser Proteinen besitzen Sortiermerkmale, die für den Weitertransport aus dem Cytosol in die Mitochondrien, Chloroplasten (bei Pflanzen), Peroxysomen oder in den Zellkern sorgen. Die meisten Proteine haben überhaupt kein spezielles Sortiermerkmal und bleiben daher ständig Bestandteile des Cytosols.
Über den zweiten Weg (ausgezogene Linie) werden Proteine befördert, die aus der Zelle ausgeschieden werden sollen, und ausserdem auch solche, deren Bestimmungsort im ER, im Golgi-Apparat, in der Plasmamembran oder in den Lysosomen liegt.
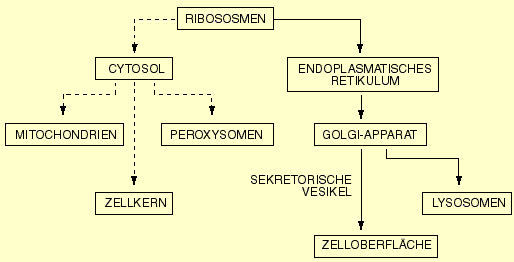
Die Prozesse, welche Proteine sortieren und zu ihrem korrekten Platz in der Zelle transportieren, bezeichnet man als Protein-Targeting (Zielsteuerung). Für Protein-Targeting sind bestimmte Sortiermerkmale nötig, welche die Proteine Schritt für Schritt durch die oben skizzierten Transportwege dirigieren. Man unterscheidet nun zwei Arten solcher Sortiermerkmale: Signalpeptide und Signalbereiche.
Signalpeptide sind kurze Aminosäuresequenzen am Aminoterminus eines neu synthetisierten Polypeptids. Diese Signalsequenzen lenken ein Protein zu seinem Bestimmungsort und werden während des Transportes oder kurz danach entfernt.
Signalbereiche sind Oberflächenstrukturen, die sich durch die Faltung des Proteins ausbilden und für das entsprechende Protein charakteristisch sind. Signalbereiche bleiben am fertigen Protein erhalten und dienen für andere Sortierschritte, z. B. für die Erkennung bestimmter Lysosomenproteine durch ein spezielles Sortierenzym im Golgi-Apparat.
Folgende Tabelle zeigt verschiedene Signalpeptide, die die einzelnen Bestimmungsorte innerhalb einer Eukaryontenzelle festlegen:
| Funktion des Signalpeptids | Signalpeptid |
|---|---|
| Transport ins ER | H3N-Met-Met-Ser-Phe-Val-Ser-Leu-Leu-Val-Gly-Ile-Phe-Trp- Ala-Thr-Glu-Ala-Glu-Gln-Leu-Thr-Lys-Cys-Glu-Val-Phe-Gln- |
| Zurückhalten im ER-Lumen | -Lys-Asp-Glu-Leu-COO- |
| Transport in die Mitochondrien | H3N-Met-Leu-Ser-Leu-Arg-Gln-Ser-Ile-Arg-Phe-Phe-Lys-Pro- Ala-Thr-Arg-Thr-Leu-Cys-Ser-Ser-Arg-Tyr-Leu-Leu- |
| Transport in den Zellkern | -Pro-Pro-Lys-Lys-Lys-Arg-Lys-Val- |
Das vielleicht am besten charakterisierte Zielsteuerungssystem in eukaryotischen Zellen beginnt am Endoplasmatischen Retikulum (ER). Die meisten lysosomalen Proteien, Membranproteine und sekretierten Proteine besitzen eine amino-terminale Signalsequenz (siehe Tabelle), die sie für einen Transport in den Innenraum des ER markiert. Diese Signalsequenz trägt wesentlich dazu bei, dass das Ribosom zum ER gelenkt wird.
Der vollständige Prozess beginnt mit der Initiation der Proteinsynthese an freien Ribosomen im Cytoplasma. Die Signalsequenz erscheint zu Beginn der Eiweissynthese, da sie sich am Aminoterminus befindet. Sobald sie aus dem Ribosom herausragt, wird sie von einem Protein-Komplex gebunden, dem Signalerkennungspartikel SRP (signal recognition particle). Durch diesen Bindungsvorgang wird die Elongation gestoppt, wenn die Signalsequenz vollständig translatiert worden ist. Das gebundene SRP steuert das Ribosom mit dem unvollständigen Polypeptid zu SRP-Rezeptoren auf der cytosolischen Seite des ER. Das entstehende Polypeptid wird an einen Peptidtranslokationskomplex im ER abgegeben, das SRP dissoziiert vom Ribosom ab, und die Synthese des Proteins wird fortgesetzt. Der Translokationskomplex transportiert das wachsende Polypeptid durch eine ATP-getriebene Reaktion in den Innenraum des ER. Dort wird die Signalsequenz von einer Signalpeptidase entfernt. Sobald das Protein zu Ende synthetisiert worden ist, dissoziiert das Ribosom vom ER ab.
Abbildung
X
Zielsteuerungssystem am ER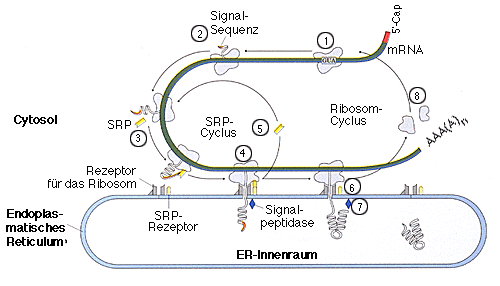
Das vielleicht am besten charakterisierte Zielsteuerungssystem beginnt am Endoplasmatischen Retikulum (ER). Die meisten lysosomalen Proteien, Membranproteine und sekretierten Proteine besitzen eine aminoterminale Signalsequenz (siehe Tabelle), die sie für einen Transport in den Innenraum des ER markiert. Diese Signalsequenz trägt wesentlich dazu bei, dass das Ribosom zum ER gelenkt wird.
Der vollständige Prozess beginnt mit der Initiation der Proteinsynthese an freien Ribosomen im Cytoplasma. Die Signalsequenz erscheint zu Beginn der Eiweissynthese, da sie sich am Aminoterminus befindet. Sobald sie aus dem Ribosom herausragt, wird sie von einem Protein-Komplex gebunden, dem Signalerkennungspartikel SRP (signal recognition particle). Durch diesen Bindungsvorgang wird die Elongation gestoppt, wenn die Signalsequenz vollständig translatiert worden ist. Das gebundene SRP steuert das Ribosom mit dem unvollständigen Polypeptid zu SRP-Rezeptoren auf der cytosolischen Seite des ER. Das entstehende Polypeptid wird an einen Peptidtranslokationskomplex im ER abgegeben, das SRP dissoziiert vom Ribosom ab, und die Synthese des Proteins wird fortgesetzt. Der Translokationskomplex transportiert das wachsende Polypeptid durch eine ATP-getriebene Reaktion in den Innenraum des ER. Dort wird die Signalsequenz von einer Signalpeptidase entfernt. Sobald das Protein zu Ende synthetisiert worden ist, dissoziiert das Ribosom vom ER ab.
Protein-Targeting bei Prokaryoten
Auch bei Bakterien findet ein Protein-Targeting zur inneren oder äusseren Membranseite oder ins extrazelluläre Medium (Sekretion) statt. Dabei werden die Signalsequenzen am Aminoterminus der Proteine verwendet, die denen der eukaryotischen Proteine, die zum ER transportiert werden, ähnlich sind.
Tabelle
X
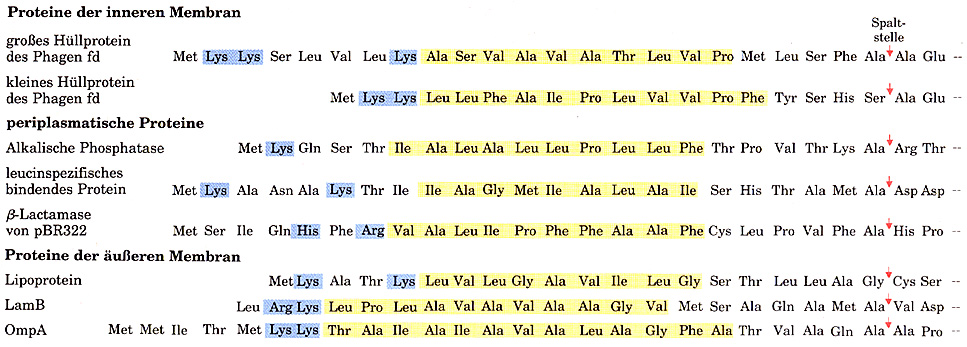
Proteine der inneren MembranSignalsequenzen, die bei Bakterien für das Targeting zu verschiedenen Orten verwendet werden. Die Spaltstellen, die das Ende der Signalsequenzen bezeichnen, sind durch Pfeile markiert.
Einige Proteine, die eine oder mehrere Membranen passieren, müssen, um ihren endgültigen Bestimmungsort zu erreichen, solange eine ganz bestimmte "translokationsfähige" Konformation einnehmen, bis dieser Vorgang abgeschlossen ist. Die funktionelle Konformation wird erst nach der Translokation eingenommen, und man findet häufig, dass Proteine, die in dieser endgültigen Form gereinigt wurden, nicht mehr transportiert werden können.
Es gibt immer mehr Hinweise darauf, dass die Translokationskonformation bei allen Bakterienzellen von einer Gruppe spezieller Proteine stabilisiert wird. Diese binden bereits an das zu transportierende Protein, während es synthetisiert wird, und hindern es daran, sich zu seiner endgültigen dreidimensionalen Struktur zu falten.
Bei E. coli fördert ein Protein, der sogenannte Trigger-Faktor, die Translokation mindestens einiger Proteine.